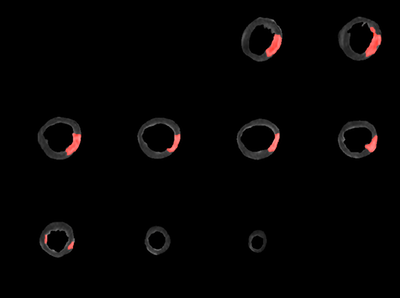
I denne øvelse skal I lave et script (dvs. ingen function header), opdelt i celler, som skal indlæse en stak af dicom billeder, som dækker hjertet på en patient med en nylig blodprop i hjertet. Billederne er dannet med en speciel MR sekvens, som selektivt fremhæver områder med forhøjet vandindhold, det såkaldte Area At Risk eller AAR. Hvis ikke blodforsyningen til dette områder genetableres med ballonudvidelse eller bypass-operation kan AAR vævet gå til grunde og blive til arvæv med tab af pumpefunktion til følge.
På samme vis som i hjerne tumor øvelsen, består øvelsen i at indlæse billederne, markere et normalt område med en ROI, udregne middel og standard deviation, og sætte pixels som overstiger m+6*sd som værende indikative for AAR. Ved at lave både en open og en close operation vises kun det afficerede område. Ved at se på længden af dette ganget med voxelstørrelsen kan størrelsen på AAR beregnes og angives.
I og med analyserne foretages på en stak af billeder, kan montage funktionen med fordel bruges til at vise resultaterne.
Instruktioner:
- Hent Hjerte.mat
- Lav en celle med flg. overskrift:
- %% Hent hjertefiler
- Hent Hjerte.mat filen med load funktionen
- Lav en celle med flg. overskrift:
- %% Indtegn normal ROI og beregn normal mean + sd
- Vis først et enkelt snit, nemlig det fjerde med funktionen imshow - husk at montage-egnede billeder er opbygget som:
- højde x bredde x 1 x snit
- Lav et roi med funktionen impoly og gem det i en variabel kaldet normalt
- Lav en maske kaldet bw med funktionen createMask med det nyskabte normalt roi
- Sæt reference vævet til at være bw .* det fjerde snit i im variablen - gem det som en variabel kaldet ref
- Find indekserne på ref og gem dem i idx variablen.
- Beregn middel og standard deviation af ref(idx) og gem dem i m og sd variablerne.
- Lav en celle med flg. overskrift:
- %% Marker væv, som overstiger threshold
- I denne celle skal de pixels, som overstiger m + 6*sd, findes og markeres som værende opladt
- Beregn myo som element produktet af im og MaskMyo
- Find idx på myo som overstiger m + 6*sd
- Sæt en variabel ved navn enh til at være en nul-matrix med størrelsen på im
- Sæt enh(idx) til 1
- Lav et string element som en disk med diameter 3. Gem den i en variabel kaldet sel
- Prøv at plotte enh med montage kommandoen - iagttag de mange forkerte opladede områder. Disse skal nu fjernes med imopen og imclose kommandoer
- Lav en imopen operation på enh for at fjerne støj. Gem outputtet i en variabel kaldet enhopen
- Lav sel om til at være en disk med diameteren 10
- Lav en imclose operation på enhopen. Gem outputtet i variablen enhclose.
- Denne operation laves for at lukke huller i enhopen
- Områderne med opladning skal markeres med rødt. Derfor skal vi have lavet en ny AARstak variabel, som har R G og B værdier, dvs. størrelsen 3 på 3. plads:
- AARstak(:,:,:,:) = repmat(myo,[1 1 3 1]);
- Nu skal pixels i enhclose markeres som værende opladte:
- myo(find(enhclose))=1;
- Sæt første farvekanal i AARstak - AARstak(:,:,1,:) - til at være lig myo variablen
- Lav en montage af AARstak
- Lav en celle med flg. overskrift:
- %% Beregn størrelsen på det afficerede væv
- Beregn Voxelsize som produktet af ResX, ResY og SlTh divideret med 1000 for at få resultatet i ml
- Beregn størrelsen af AAR som produktet af Voxelsize og længden af enhclose variablen
- Sæt titlen på billedet til:
- title(sprintf('Area At Risk (AAR) = %2.2f ml',AAR));
- Gem scriptet som HjerteMontage.m